Varios grupos del CIBER-BBN en la Universidad de Barcelona, el Instituto de Química Avanzada de Cataluña (IQAC-CSIC), el Instituto de Microelectrónica de Barcelona (IMB-CNM-CSIC) y el Instituto de Nanociencia y Materiales de Aragón (INMA) —un instituto mixto del CSIC y de la Universidad de Zaragoza (UNIZAR)— han desarrollado un nuevo método para detectar virus ARN basado en la tecnología de uso de sondas formadoras de estructuras tríplex. Esta metodología innovadora abre nuevas opciones para detectar virus como el SARS-CoV-2, el virus de la gripe A (H1N1) o el virus sincitial respiratorio (VSR), un patógeno que afecta a recién nacidos y que exige un cuidado diagnóstico diferencial.
Este trabajo interdisciplinario, publicado en la revista International Journal of Molecular Sciences, está liderado por Carlos J. Ciudad y Verónica Noé, de la Facultad de Farmacia y Ciencias de la Alimentación y del Instituto de Nanociencia y Nanotecnología (IN2UB) de la Universidad de Barcelona; Ramón Eritja, Anna Aviñó, Lluïsa Vilaplana y M.Pilar Marco, del IQAC-CSIC y el CIBER-BBN; Manuel Gutiérrez, Antoni Baldi y César Fernández, del IMB-CNM-CSIC, y Valeria Grazu y Jesús Martínez, investigadores del CSIC en el Instituto de Nanociencia y Materiales y de Aragón INMA (CSIC-UNIZAR) y del CIBER-BBN.
Este trabajo de investigación se realizó en el contexto del proyecto PoC4CoV, liderado por M. Pilar Marco y César Fernández y financiando a través de la Plataforma Temática Interdisciplinar de Salud Global del CSIC. Posteriormente, la investigación ha continuado como parte de un proyecto financiado por La Marató de TV3 de 2020 para luchar contra la COVID-19 en el que también participan expertos de la Facultad de Química de la UB.
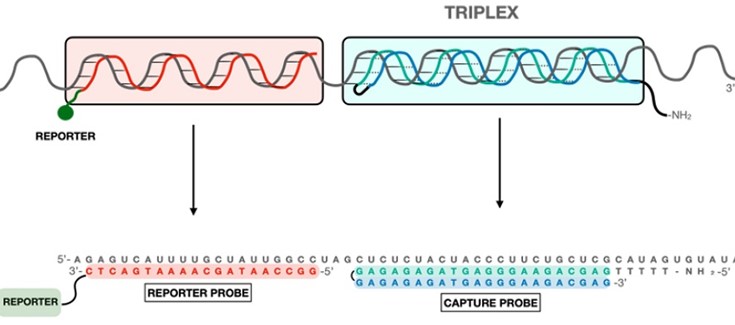
La nueva metodología se basa en la capacidad de las pinzas de polipurinas (PPRHs) —diseñadas por el grupo de terapia del cáncer de la UB— para capturar el ARN viral y formar un tríplex de alta afinidad. Cuando esta estructura híbrida se conecta con una sonda molecular y se pone en contacto con la muestra del paciente afectado, se obtiene una señal de detección del agente viral. El método presentado ahora en la publicación científica se ha denominado Triplex Enhanced Nucleic Acid Detection Assay (TENADA).
«Las PPRHs son horquillas de ADN de una sola cadena no modificado que están formadas por dos dominios especulares de polipurinas antiparalelas. Estos dominios, conectados entre sí por un bucle de timidina, se unen por enlaces intramoleculares de Hoogsteen inversos. Las pinzas moleculares pueden unirse de forma específica a secuencias de polipirimidinas en virus de ADN monocatenario (ADNmc), bicatenario (ADNbc) o ARN mediante enlaces de Watson-Crick, formando así un tríplex antiparalelo», detalla el catedrático Carlos J. Ciudad, del Departamento de Bioquímica y Fisiología de la UB.
Entre las ventajas que presenta en la detección del ARN vírico, cabe destacar que la metodología de las PPRHs puede aplicarse sin la intervención de la transcriptasa inversa —la enzima que convierte el ARN en ADN— ni del termociclador (el aparato que amplifica las muestras material genético con la reacción en cadena de la polimerasa o PCR). Además, tiene una sensibilidad y especificidad equivalente a la de la prueba PCR y puede aportar resultados en menos de una hora.
En el marco del trabajo, el equipo utilizó el formato sándwich de hibridación en varios dispositivos de biodetección. Esta estrategia utiliza dos oligonucleótidos: una horquilla de PPRH formadora de tríplex que actúa como sonda de captura y un oligonucleótido de ADN formador de dúplex marcado que actúa como sonda de detección.
«Las horquillas de PPRH que forman tríplex se diseñaron para unirse a secuencias de polipirimidina del SARS-CoV-2, mientras que las sondas de detección se diseñaron como complementarias de una región cercana al lugar de la diana de las polipirimidinas. Así, la presencia del ARN del SARS-CoV-2 se detecta mediante la formación del complejo ternario en la superficie del biosensor», detalla la catedrática Verónica Noé (UB-IN2UB).
Esta metodología se ha implementado en un dispositivo electroquímico compacto que integra una celda electroquímica de dos electrodos en un chip —fabricada en la Sala Blanca de Micro y Nanofabricación del IMB-CNM-CSIC— y un componente fluídico en papel, y en un sistema de flujo lateral térmico implementado en nitrocelulosa y utilizando nanopartículas plasmónicas y papel térmico que se ha desarrollado en el INMA (CSIC-UNIZAR).
Las PPRHs están descritas en la bibliografía científica como herramientas de silenciamiento génico de diversos genes implicados principalmente en el cáncer. Además, también se han incorporado como sondas en biosensores para la detección de pequeñas moléculas de ARN (micro-ARN) para determinar el estado de metilación del ADN y para el diagnóstico de neumonía causada por el hongo Pneumocystis jirovecii.
Ahora, la nueva metodología TENADA se revela eficaz no sólo en la detección de partículas virales. La alta afinidad de las PPRHs por el ARN viral es una propiedad que puede aplicarse para inhibir el proceso de replicación del virus. Por este motivo, ahora también son objeto de estudio las propiedades antivirales de las pinzas de polipurinas CC1PPRH y CC2PPRH en células del linaje VeroE6 infectadas con viriones SARS-CoV-2.
En paralelo, el trabajo de los distintos grupos implicados también ha sido la base de una tecnología que se patentó y licenció en julio de 2022 mediante la participación del Centro de Patentes de la UB, el CSIC y el CIBER-BBN. A su vez, esta patente ha sido licenciada de manera no exclusiva a la empresa española Nanoinmunotech a través de la gestión de la Fundación Bosch i Gimpera (FBG-UB) en el proceso de protección de la tecnología y el acuerdo de licencia de la empresa.
Artículo de referencia:
Aviñó, A.; Cuestas-Ayllón, C.; Gutiérrez-Capitán, M.; Vilaplana, L.; Grazu, V.; Noé, V.; Balada, E.; Baldi, A.; Félix, A.J.; Aubets, E.; Valiuska, S.; Domínguez, A.; Gargallo, R.; Eritja, R.; Marco, M.-P.; Fernández-Sánchez, C.; Martínez de la Fuente, J.; Ciudad, C.J. «Detection of SARS-CoV-2 Virus by Triplex Enhanced Nucleic Acid Detection Assay (TENADA)». International Journal of Molecular Sciences, diciembre de 2022. Doi:10.3390/ijms232315258