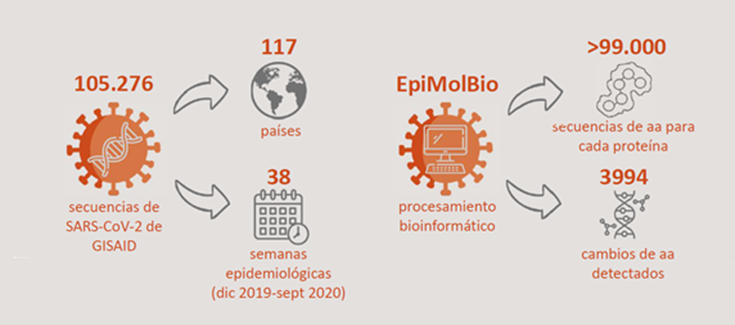
El equipo de la investigadora del CIBERESP África Holguín en el Hospital Ramón y Cajal-IRYCIS de Madrid, integrado en el grupo que lidera Juan Carlos Galán, ha llevado a cabo un estudio sobre la diversidad genética del SARS-CoV-2, crucial para comprender su evolución y evaluar su impacto en el diagnóstico, vacunas y terapias contra la COVID-19. Para ello se analizaron datos de más de 100.000 aislados de SARS-CoV-2 de 117 países.
El trabajo ha sido publicado en la revista Viruses y analiza el grado de conservación de aminoácidos y la evolución temporal a nivel global y regional por semana epidemiológica en cada residuo de las cuatro proteínas estructurales del SARS-CoV-2: espícula, membrana, envuelta y nucleocápside. El análisis fue llevado a cabo utilizando la herramienta bioinformática EpiMolBio, desarrollada en el propio grupo investigador.
En las 105,276 secuencias virales disponibles, los autores observaron que, a pesar de la conservación extremadamente alta de las proteínas estructurales del SARS-CoV-2 (> 99%), todas presentaron cambios de aminoácido con diferentes tendencias temporales. Así, encontraron 142 cambios en 65 de los 75 aminoácidos de la envuelta, 291 cambios en 165 de los 222 residuos de la membrana, 890 cambios en 359 de los 419 residuos de la nucleocápside, y 2.671 cambios en 1.132 de los 1.273 residuos de la espícula.
La evolución de los cambios difirió por regiones geográficas y por semana epidemiológica, siendo los más prevalentes D614G (81,5%) en la proteína de la espícula, seguido de la combinación R203K + G204R (37%) en la proteína de la nucleocápside. La proteína de la espícula albergaba la mayor proporción de cambios a lo largo de su secuencia (88,9% de 1273 aa), seguida de la proteína de la envuelta (86,6% de 75 aa), la proteína de la nucleocápside (85,7% de 419 aa) y la proteína de la membrana (74,3% de 222 aa).
"Estos datos aportan información extremadamente útil para enfoques futuros de diagnóstico, tratamiento, eficacia de vacunación o diseño de vacunas dirigidas frente a esas proteínas estructurales del SARS-CoV-2 y será de gran relevancia en los esfuerzos de prevención y control de la enfermedad de la COVID-19, y para comprender mejor cómo se está propagando este virus entre los distintos países" concluye la Dra. Holguín.
La primera autora del trabajo, Paloma Troyano, tiene un contrato de Investigación Rio Hortega y el segundo autor, Roberto Reinosa, es colaborador bioinformático para el desarrollo de la herramienta EpimolBio.
Artículo de referencia:
Evolution of SARS-CoV-2 Envelope, Membrane, Nucleocapsid, and Spike structural proteins from the beginning of the pandemic to September 2020: a global and regional approach by epidemiological week. Paloma Troyano-Hernáez, Roberto Reinosa and África Holguín. Viruses 2021, 13(2), 243. https://www.mdpi.com/1999-4915/13/2/243